Was sind Exons?
- Ein Exon ist jeder Teil eines Gens, der einen Teil der endgültigen reifen RNA bildet, nachdem Introns durch RNA-Spleißen entfernt wurden.
- Exons beziehen sich sowohl auf die DNA-Sequenz innerhalb eines Gens als auch auf die entsprechende Sequenz in RNA-Transkripten.
- Beim RNA-Spleißen werden Introns entfernt und Exons kovalent verbunden, um reife RNA zu erzeugen.
- Der Begriff „Exon“ wurde 1978 von Walter Gilbert geprägt und leitet sich von der exprimierten Region eines Gens ab.
- Ursprünglich wurden Exons für proteinkodierende Transkripte definiert, die vor der Translation gespleißt werden.
- Zu den Exons gehören auch Sequenzen, die aus rRNA, tRNA und anderen nichtkodierenden RNA-Molekülen entfernt wurden.
- Exons kodieren Teile des Endprodukts eines Gens, beispielsweise Proteine, in eukaryotischen Genen.
- Exons kommen ausschließlich in eukaryotischen Genen vor.
- Exons sind die kodierenden Regionen eines Gens und wichtige Segmente.
- Introns, nichtkodierende Regionen, Interspace-Exons in einem Gen.
- Beim Spleißen, einem posttranskriptionellen Prozess, werden Introns entfernt und Exons verbunden.
- Exons enthalten die Informationen, die zur Kodierung von Proteinen erforderlich sind.
- Gene in Eukaryoten bestehen aus kodierenden Exons, die mit nicht-kodierenden Introns durchsetzt sind.
- Introns werden entfernt, um eine funktionelle Boten-RNA (mRNA) für die Proteintranslation zu erzeugen.
Die Geschichte des Begriffs „Exon“ reicht bis ins Jahr 1978 zurück, als Walter Gilbert, ein amerikanischer Biochemiker, das Konzept einführte. Gilbert schlug eine neue Perspektive auf die Struktur von Genen vor und schlug vor, dass die traditionelle Idee eines Cistrons durch eine Transkriptionseinheit ersetzt werden sollte, die aus abwechselnden Regionen besteht, die von der reifen Boten-RNA (mRNA) verloren gehen würden, die er als Introns bezeichnete mit Regionen, die ausgedrückt werden würden, die er Exons nannte.
Ursprünglich wurde diese Definition von Exons speziell auf proteinkodierende Transkripte angewendet, die vor der Translation gespleißt werden. Der Fokus lag auf den Genregionen, die in reife mRNA transkribiert und schließlich in Proteine übersetzt werden würden.
Im Laufe der Zeit wurde die Definition von Exons auf andere Arten von RNA-Molekülen ausgeweitet. Sequenzen, die aus ribosomaler RNA (rRNA), Transfer-RNA (tRNA) und anderer nicht-kodierender RNA (ncRNA) entfernt wurden, wurden auch als Exons bezeichnet. Diese Ausweitung des Begriffs umfasste ein breiteres Spektrum von RNA-Molekülen, die aus verschiedenen Teilen des Genoms stammen.
Darüber hinaus wurde das Konzept der Exons weiterentwickelt und umfasste auch das Transspleißen, einen Prozess, bei dem RNA-Moleküle aus verschiedenen Genomregionen miteinander verbunden werden. In diesem Zusammenhang bezeichneten Exons die Segmente von RNA-Molekülen, die aus verschiedenen Teilen des Genoms stammen und durch Transspleißen verbunden wurden.
Insgesamt hat der Begriff „Exon“ seit seiner Einführung eine Erweiterung und Verfeinerung erfahren, beginnend mit seiner Anwendung auf proteinkodierende Transkripte und später mit der Einbeziehung anderer Arten von RNA-Molekülen und Transspleißvorgängen. Dieses sich weiterentwickelnde Verständnis von Exons hat zu unserem Wissen über die Genstruktur und die RNA-Verarbeitung in verschiedenen biologischen Kontexten beigetragen.
Exon-Definition
Exon: Eine kodierende Region eines Gens, die die zur Produktion eines Proteins erforderlichen Informationen enthält oder in die RNA-Struktur eingebaut ist.
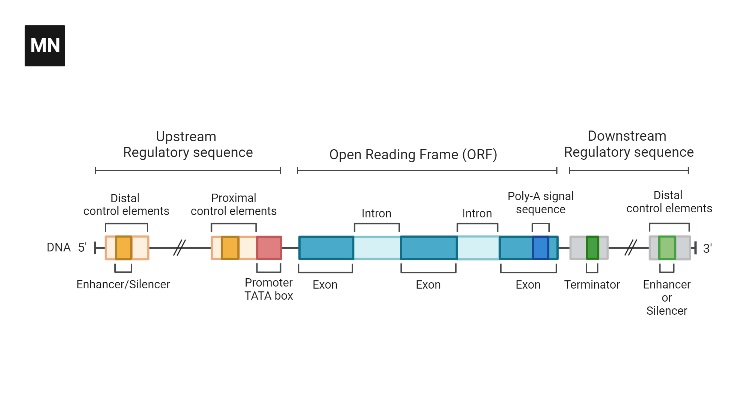
Struktur von Exon
- Exons sind Nukleotidsequenzen in DNA oder RNA, die exprimiert werden.
- Exons sind sowohl in der DNA als auch in der reifen RNA konserviert.
- Die Exonstruktur umfasst sowohl die kodierende Sequenz als auch die nicht translatierten Regionen (UTRs) vom 5′- und 3′-Ende.
- In proteinkodierenden Genen enthält das erste Exon oft sowohl die 5′-UTR als auch den ersten Teil der kodierenden Sequenz, es können aber auch Exons vorkommen, die ausschließlich aus UTR-Regionen oder 3′-UTR-Regionen bestehen.
- Nichtkodierende RNA-Transkripte können auch Exons und Introns enthalten.
- Alternatives Spleißen kann dazu führen, dass verschiedene reife mRNA-Moleküle, die aus demselben Gen stammen, unterschiedliche Sätze von Exons haben, da unterschiedliche Introns in der Prä-mRNA entfernt werden können.
- Unter Exonisierung versteht man die Schaffung neuer Exons durch Mutationen in Introns.
- Exons bestehen aus DNA-Abschnitten, die in Aminosäuren und Proteine übersetzt werden.
- In eukaryontischen Organismen können Exons innerhalb eines Gens zusammenhängend oder in einem diskontinuierlichen Gen durch Introns getrennt sein.
- Bei der Transkription wird das Gen in Prä-mRNA umgeschrieben, die sowohl Introns als auch Exons enthält.
- Die Prä-mRNA wird einer Verarbeitung unterzogen, bei der Introns herausgespleißt werden, was zu reifer mRNA führt.
- Reife mRNA besteht aus Exons und kurzen untranslatierten Regionen (UTRs) an beiden Enden.
- Die Exons bilden den endgültigen Leserahmen, der aus in Tripletts angeordneten Nukleotiden besteht.
- Der Leserahmen beginnt mit einem Startcodon (normalerweise AUG) und endet mit einem Terminationscodon.
- Die Nukleotide im Leserahmen sind in Tripletts angeordnet, wobei jedes Triplett für eine Aminosäure kodiert.
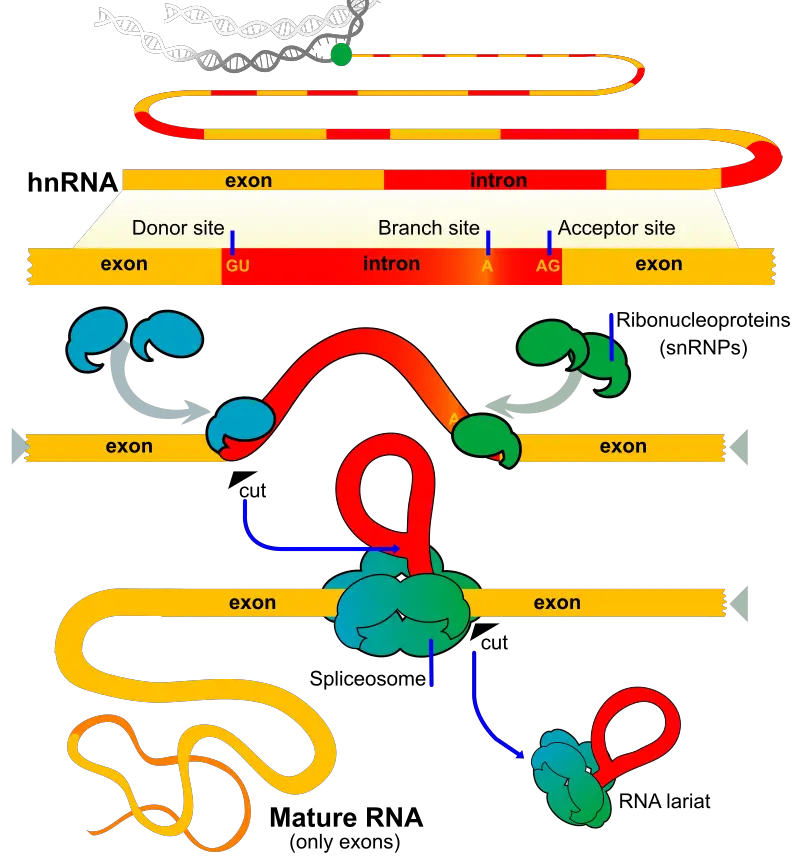
Was ist Spleißen?
Das Spleißen ist ein entscheidender Prozess in der Genexpression, bei dem Introns, die nicht-kodierenden Regionen, aus dem primären mRNA-Transkript entfernt und Exons zusammengefügt werden, um ein reifes mRNA-Molekül zu bilden. Hier sind einige wichtige Punkte zum Spleißen:
- Bildung reifer mRNA: Durch das Spleißen wird die anfängliche Prä-mRNA, die sowohl Exons als auch Introns enthält, in ein reifes mRNA-Molekül umgewandelt, das nur die kodierenden Exons enthält. Diese reife mRNA dient als Vorlage für die Proteinsynthese.
- Tritt vor der Übersetzung auf: Das Spleißen findet vor dem Translationsprozess statt, bei dem es sich um die Synthese von Proteinen handelt. Es stellt sicher, dass die richtige genetische Information in der mRNA für eine ordnungsgemäße Proteinproduktion erhalten bleibt.
- Bedeutung für die Proteinkodierung: Das Spleißen ist für die genaue Kodierung von Proteinen unerlässlich. Ohne Spleißen würden die Protein-kodierenden Regionen durch nicht-kodierende Introns unterbrochen, was zur Produktion nicht-funktionaler oder verkürzter Proteine führen würde.
- Rolle bei der Genexpressionsregulation: Das Spleißen spielt eine wichtige Rolle bei der Regulierung der Genexpression. Alternatives Spleißen ermöglicht die Produktion verschiedener mRNA-Isoformen aus einem einzigen Gen, was zur Synthese verschiedener Proteinvarianten mit unterschiedlichen Funktionen führt. Dies trägt zur Komplexität und Vielfalt der Genexpression bei.
- Einflussfaktoren auf das Spleißen: Das Spleißen kann je nach Art des Organismus, der RNA- oder Intronstruktur und dem Vorhandensein spezifischer Katalysatoren variieren. Verschiedene Organismen und Zelltypen können unterschiedliche Spleißmuster und -mechanismen aufweisen.
- Spleißmethoden: Es wurden mehrere Mechanismen und Wege zum Spleißen identifiziert, darunter der Hauptspleißosomenweg und alternative Spleißwege. Diese Mechanismen umfassen die präzise Erkennung und Entfernung von Introns und die präzise Verbindung von Exons, um die reife mRNA zu erzeugen.
Spleißen Prozess
Beim Spleißen werden Introns aus der Prä-mRNA erkannt und entfernt, um reife mRNA zu erzeugen. Hier sind die wichtigsten Schritte beim Spleißen:
- Konservierte Sequenzen: Die Prä-mRNA enthält Exon-Intron-Verbindungen mit konservierten Sequenzen. Die 5'-Endverbindung hat eine GU-Sequenz, die als 5'-Spleißstelle bekannt ist, während die 3'-Endverbindung eine AG-Sequenz hat, die als 3'-Spleißstelle bezeichnet wird.
- Standort der Abzweigstelle: Stromaufwärts der 3′-Spleißstelle befindet sich eine invariante Stelle, die 15–45 Nukleotide entfernt liegt. Diese Stelle ist reich an Adenin (A)-Basen und wird als Verzweigungspunkt oder Verzweigungsstelle bezeichnet.
- Erkennung von Spleißstellen: Das Spleißosom, ein Komplex aus RNA- und Proteinkomponenten, erkennt diese konservierten Sequenzen in der Prä-mRNA an den jeweiligen Spleißstellen. Das Spleißosom besteht aus kleinen nuklearen Ribonukleoproteinen (snRNPs), die aus kleiner nuklearer RNA (snRNA) und zugehörigen Proteinen bestehen.
- Zusammensetzung des Spleißosoms: Die wichtigsten snRNPs, die an der Spleißosomenbildung beteiligt sind, sind U1, U2, U5 und der U4-U6-Komplex. U4 und U6 sind innerhalb des Spleißosoms als eine Einheit miteinander verbunden.
- Spleißosomenmontage: Das Spleißosom setzt sich auf der Prä-mRNA zusammen und bringt die snRNPs und andere assoziierte Proteine an den Spleißstellen zusammen. Diese komplexe Formation ermöglicht eine präzise Erkennung und Bindung der Spleißstellen.
- Spleißosomenkatalyse: Das Spleißosom katalysiert zwei Umesterungsreaktionen. Die erste Reaktion beinhaltet die Spaltung an der 5‘-Spleißstelle, was zur Bildung eines freien 5‘-Endes des Introns und einer mit dem Verzweigungspunkt verbundenen Intron-Lariat-Struktur führt. In der zweiten Reaktion wird das Intron freigesetzt und die benachbarten Exons werden miteinander verbunden, sodass ein zusammenhängendes mRNA-Molekül entsteht.
- Exon-Ligation: Die Exons werden durch das Spleißosom miteinander verbunden und das Intron wird als Lassostruktur freigesetzt. Die resultierende reife mRNA enthält nur die kodierenden Exons und ist bereit für die Translation in ein Protein.
Insgesamt umfasst der Prozess des Spleißens die Erkennung konservierter Sequenzen an den Exon-Intron-Übergängen, den Zusammenbau des Spleißosoms, die Katalyse der Spleißreaktionen und die Ligation von Exons, um reife mRNA zu erzeugen. Dieser präzise und regulierte Prozess gewährleistet die genaue Entfernung von Introns und die ordnungsgemäße Verbindung von Exons, was zur Produktion funktioneller mRNA für die Proteinsynthese führt.
Spleißpfad
Der Spleißweg umfasst eine Reihe von Schritten, die durch kleine nukleare Ribonukleoproteine (snRNPs) erleichtert werden, um Introns zu entfernen und Exons in prä-mRNA zu verbinden. Hier ist eine Übersicht über den Spleißpfad:
- U1- und U2-Bindung: U1-snRNP bindet an die 5′-Spleißstelle und U2-snRNP bindet an die invariante Stelle oder den Verzweigungspunkt innerhalb des Introns.
- Bindung zusätzlicher snRNPs: Andere snRNPs, darunter U5 und U4-U6, binden an die Intronregion und führen so zum Aufbau des Spleißosomenkomplexes.
- Spleißosomenmontage: Der Spleißosomenkomplex entsteht durch die Bindung von snRNPs und assoziierten Proteinen. Dieser Komplex bringt die Intron- und Exon-Sequenzen in unmittelbare Nähe.
- Schleifen und Intronentfernung: Das Spleißosom führt eine Schleife aus dem Intron aus und bringt die beiden Enden des Introns nahe aneinander.
- Freisetzung und Neuordnung von snRNPs: U1- und U4-snRNPs werden vom Spleißosom freigesetzt, während U6-snRNP sowohl an die 5′-Spleißstelle als auch an U2-snRNP gebunden ist.
- Spaltung and ligation: Das 5′-Ende des Introns wird abgespalten und das abgespaltene Ende bindet an den Verzweigungspunkt, der reich an Adenin (A) ist. Anschließend wird das 3‘-Ende des Introns abgespalten, was zur Freisetzung des Introns in einer schleifenartigen Struktur namens Lariat führt.
- Exon-Beitritt: Die beiden benachbarten Exons werden miteinander verbunden und bilden ein kontinuierliches reifes mRNA-Molekül.
- Fortsetzung des Spleißens: Die snRNPs, die am Spleißen eines Introns beteiligt waren, werden freigesetzt und können für das Spleißen anderer Introns innerhalb der prä-mRNA wiederverwendet werden. Der Spleißvorgang wird fortgesetzt, bis alle Introns entfernt wurden.
- Intron-Abbau: Das freigesetzte Intron wird in seiner Lariat-Form durch Enzyme weiter abgebaut und seine Bestandteile können für andere zelluläre Prozesse recycelt werden.
Indem er diesem Spleißweg folgt, sorgt der Spleißosomenkomplex für die präzise Entfernung von Introns und die genaue Verbindung von Exons, was zur Erzeugung reifer mRNA führt, die in Proteine übersetzt werden kann.
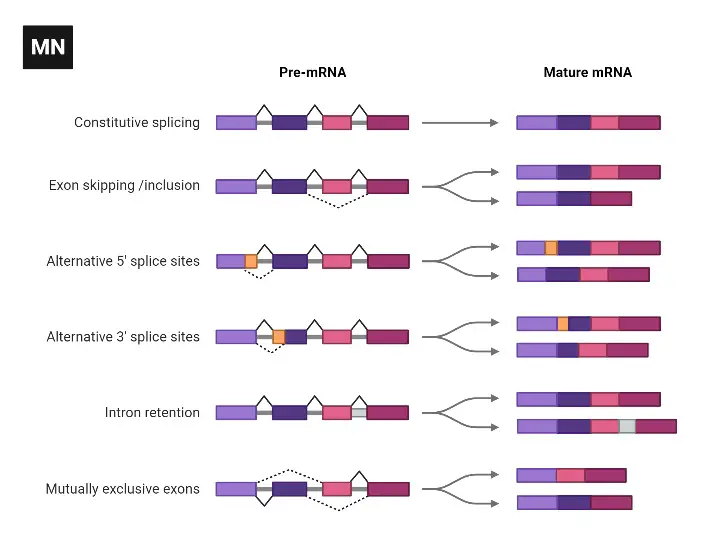
Alternatives Spleißen
Alternatives Spleißen ist ein Mechanismus, der die Produktion verschiedener mRNA-Isoformen durch selektives Zusammenfügen verschiedener Exons während der RNA-Verarbeitung ermöglicht. Dieser Prozess führt zur Erzeugung mehrerer Proteinvarianten aus einem einzigen Gen. Hier sind die wichtigsten Arten des alternativen Spleißens:
- Aufeinanderfolgendes Spleißen: Bei dieser Art des alternativen Spleißens werden aufeinanderfolgende Introns herausgespleißt und die benachbarten Exons miteinander verbunden. Dies führt zum Einschluss bestimmter Exons und zum Ausschluss anderer, wodurch mRNA-Varianten mit unterschiedlichen Exon-Zusammensetzungen entstehen.
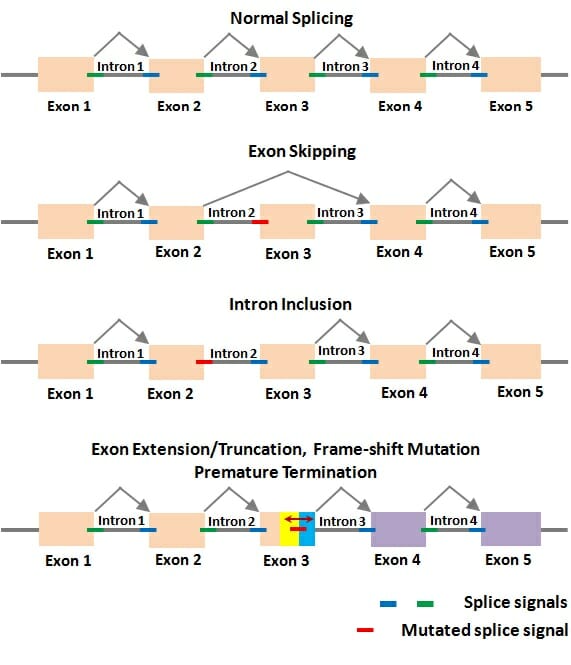
- Exon-Skipping: Beim Exon-Skipping werden bestimmte Exons zusammen mit ihren angrenzenden Exons vor der Translation aus der Prä-mRNA herausgeschnitten. Dies führt zum Ausschluss spezifischer Exons aus der endgültigen mRNA, was zur Produktion von Proteinisoformen führt, denen diese Exons fehlen.
- Alternative 5′-Spleißstelle oder 3′-Spleißstelle: Alternatives Spleißen kann auch durch die Verwendung alternativer 5′- oder 3′-Spleißstellen erfolgen. Dies bedeutet, dass verschiedene Exons an alternativen Spleißstellen zusammengefügt werden können, was zu mRNA-Isoformen mit unterschiedlichen Exon-Einschlussmustern führt.
- Intron-Retention: In einigen Fällen bleiben bestimmte Introns in der reifen mRNA erhalten, anstatt herausgespleißt zu werden. Dies führt zum Einbau intronischer Sequenzen in die mRNA und kann funktionelle Konsequenzen für die resultierende Proteinisoform haben.
Das Phänomen des alternativen Spleißens erhöht die Vielfalt der Proteine, die aus einer begrenzten Anzahl von Genen erzeugt werden können, erheblich. Durch selektives Einschließen oder Ausschließen von Exons kann alternatives Spleißen die kodierende Sequenz und damit die Struktur, Funktion und Regulierung von Proteinen verändern. Es spielt eine entscheidende Rolle bei der gewebespezifischen Genexpression, Entwicklungsprozessen und der Erzeugung von Proteinvielfalt in komplexen Organismen.
Funktion von Exon
- Exons kodieren Proteine: Exons sind Abschnitte der kodierenden DNA, die die für die Produktion von Proteinen notwendigen Informationen enthalten. Verschiedene Exons innerhalb eines Gens können unterschiedliche Domänen eines Proteins kodieren.
- Bildung von Proteindomänen: Exons können Proteindomänen einzeln kodieren oder zusammengefügt werden, um funktionelle Proteindomänen zu bilden. Dieses Spleißen von Exons ermöglicht die Schaffung verschiedener Proteinstrukturen und -funktionen.
- Molekulare Evolution durch Exon-Shuffling: Das Vorhandensein von Exons und Introns ermöglicht die molekulare Evolution durch einen Prozess namens Exon-Shuffling. Exon-Shuffling tritt auf, wenn Exons während der Rekombination zwischen Schwesterchromosomen ausgetauscht werden, was zur Bildung neuer Gene mit unterschiedlichen Exon-Kombinationen führt.
- Alternatives Spleißen: Exons spielen auch eine entscheidende Rolle beim alternativen Spleißen, einem Prozess, der die Produktion mehrerer Proteinisoformen aus einem einzigen Gen ermöglicht. Alternatives Spleißen beinhaltet unterschiedliche Anordnungen von Exons, wenn Introns während der RNA-Verarbeitung entfernt werden. Dies kann den vollständigen Ausschluss eines Exons, den teilweisen Einschluss eines Exons oder sogar den Einschluss von Teilen von Introns umfassen.
- Erzeugung von Proteinvielfalt: Alternatives Spleißen ermöglicht die Erzeugung mehrerer Proteinvarianten aus einem einzigen Gen, wodurch die Proteinvielfalt und die funktionelle Komplexität erhöht werden. Alternatives Spleißen kann am selben Ort erfolgen, um unterschiedliche Isoformen mit ähnlichen Rollen oder in unterschiedlichen Zell- oder Gewebetypen zu erzeugen, wodurch das Spektrum der Proteinfunktionen erweitert wird.
- Auswirkungen auf Krankheiten: Alternatives Spleißen sowie Defekte beim alternativen Spleißen können zu verschiedenen Krankheiten führen. Erkrankungen wie Alkoholismus und Krebs wurden mit Anomalien beim alternativen Spleißen in Verbindung gebracht, was die Bedeutung einer ordnungsgemäß regulierten Exon-Nutzung für die Aufrechterhaltung einer normalen Zellfunktion unterstreicht.

FAQ
Was ist ein Exon?
Ein Exon ist ein DNA- oder RNA-Segment, das kodierende Sequenzen enthält und an der Produktion reifer RNA-Moleküle beteiligt ist.
Welche Rolle spielen Exons bei der Genexpression?
Exons spielen eine entscheidende Rolle bei der Genexpression, indem sie die Sequenzen kodieren, die in Proteine übersetzt werden. Sie sind für die kodierenden Regionen eines Gens verantwortlich.
Wie unterscheiden sich Exons und Introns?
Exons sind kodierende Regionen eines Gens, die Informationen für die Proteinsynthese enthalten, während Introns nicht kodierende Regionen sind, die zwischen Exons verstreut sind.
Wie hängen Exons und Introns mit dem RNA-Spleißen zusammen?
Beim RNA-Spleißen werden Introns aus der Prä-mRNA entfernt und Exons zusammengefügt, um reife mRNA zu bilden, die in Proteine übersetzt werden kann.
Können Exons für verschiedene Domänen eines Proteins kodieren?
Ja, Exons können für verschiedene Domänen eines Proteins kodieren. Verschiedene Exons können in verschiedenen Kombinationen zusammengefügt werden, um Proteinisoformen mit unterschiedlichen funktionellen Domänen zu erzeugen.
Was ist alternatives Spleißen und in welcher Beziehung steht es zu Exons?
Alternatives Spleißen ist ein Prozess, bei dem verschiedene Kombinationen von Exons selektiv miteinander verbunden werden, was zur Produktion mehrerer mRNA-Isoformen aus einem einzelnen Gen führt. Alternatives Spleißen erhöht die Proteinvielfalt.
Wie tragen Exons zur Proteinvielfalt bei?
Exons tragen durch alternatives Spleißen zur Proteinvielfalt bei. Durch Einschluss oder Ausschluss spezifischer Exons können aus einem einzelnen Gen unterschiedliche Proteinisoformen mit jeweils einzigartigen funktionellen Eigenschaften erzeugt werden.
Sind Exons über verschiedene Arten hinweg konserviert?
Exons weisen im Allgemeinen eine höhere Konservierung auf als Introns. Kodierungssequenzen, die für die Proteinsynthese verantwortlich sind, sind aufgrund ihrer funktionellen Bedeutung bei verschiedenen Spezies tendenziell stärker konserviert.
Können Mutationen in Exons auftreten?
Ja, Mutationen können in Exons auftreten. Mutationen in Exons können zu Veränderungen in der Aminosäuresequenz von Proteinen führen, die möglicherweise die Proteinfunktion beeinträchtigen und zu genetischen Störungen oder Krankheiten führen.
Welchen Einfluss haben Exons auf die Genregulation und die Proteinfunktion?
Exons können die Genregulation beeinflussen, indem sie regulatorische Elemente wie Enhancer oder Silencer enthalten. Darüber hinaus können verschiedene Exons Proteinisoformen mit unterschiedlichen Strukturen oder funktionellen Domänen produzieren, die sich auf die Proteinfunktion und zelluläre Prozesse auswirken.
Bibliographie
- https://www.genome.gov/genetics-glossary/Exon
- https://en.wikipedia.org/wiki/Exon
- https://biologydictionary.net/exon/
- https://www.news-medical.net/life-sciences/What-are-introns-and-exons.aspx
- https://www.biologyonline.com/dictionary/exon
- https://www.aatbio.com/resources/faq-frequently-asked-questions/What-is-exon-shuffling
